科研进展
利用家鸡二代测序数据的结构变异鉴定工具的综合评价
结构变异(structural variation,SV)指基因组中长度超过50bp的变异,通常包括缺失(DEL)、插入(INS)、重复(DUP)、倒置(INV)和易位(BND),其中DEL和DUP也被称为拷贝数变异(CNVs)。越来越多的研究表明,SV在人体进化与疾病中发挥着重要作用,同时在家畜和植物的驯化及培育过程中也扮演着关键角色。例如,在家鸡中,17.7kb的DEL和4.2kb的INS分别导致鸡脚生羽和蓝色蛋壳表型。因此,鉴定SV对基因组解释和功能验证至关重要。随着全基因组测序(WGS)数据的迅速增长,出现了大量的结构变异检测工具。研究表明,不同类型和大小的SV在模拟数据与真实数据中的检测能力和准确性存在差异。至今,没有单一的SV检测工具能够稳定地检测所有类型的SV。因此,一些研究利用多种检测工具的组合并获取重叠结果以减少假阳性率。尽管SV检测工具已被广泛使用,目前仍缺乏已验证的SV和群体WGS数据来综合评价其检测性能和适用性。
中国科学院昆明动物所张亚平院士团队利用家鸡基因组重测序数据,对十个热门结构变异检测工具在五种常见变异的检测性能进行了全面评估。结果显示,四个SV检测工具(GRIDSS、Lumpy、Wham、Manta)表现出更高的准确性。不同SV检测工具之间存在显著的异质性,且没有一个工具能够适用于所有类型和长度范围的SV。本研究建议在二代数据的SV检测中不应采用多种检测工具的组合使用策略,同时强调真实数据集和验证的SV对于评估现有及未开发的SV检测工具的性能至关重要。
该研究成果以“Comprehensive evaluation and guidance of structural variation detection tools in chicken whole genome sequence data”为题发表于生物学国际期刊《BMC genomic》上 (https://doi.org/10.1186/s12864-024-10875-1)。中国科学院昆明动物所博士毕业生马成(现于瑞典乌普萨拉大学)、博士研究生施贤和云南农业大学博士研究生李旭真为本文共同第一作者;昆明动物所张亚平院士、彭旻晟研究员共同通讯作者。该研究得到中国科学院第二次青藏高原科考专项、国家自然科学基金、云南省科技厅、春城计划、兴滇英才支持计划项目的资助以及中国西南野生生物种质资源库动物种质资源库的支持。


图 1不同工具的对不同类型的SVs的检测一致性评估 (A) INS, (B) DUP, (C) INV以及(D) DEL.
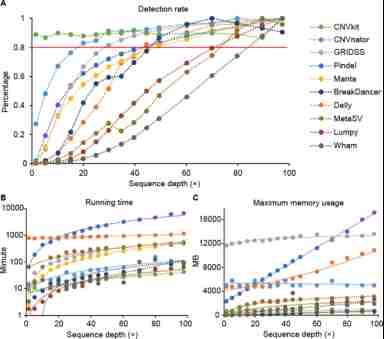
图 2测序深度对SVs检测工具的检测性能和结果的影响
发布者:施贤
发布日期:2024-10-29
来源:分子进化与基因组多样性学科组
链接:https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-024-10875-1#citeas
